Dispositif d'enregistrement de l'histoire cellulaire
Les chercheurs de l'ETH développent, à l'aide du système Crispr/Cas, un nouveau mécanisme de stockage qui produit des fragments d'ADN fournissant des informations sur certains processus cellulaires. Cette mémoire cellulaire pourra peut-être même être utilisée à l'avenir pour des diagnostics.
Les attaques virales, le contact avec des toxines environnementales ou le stress de la faim laissent des traces moléculaires dans les cellules : de tels événements modifient l'activité des gènes, ce qui se reflète dans l'ARN messager (ARNm). Les gènes y sont traduits lorsqu'ils sont activés et lus lors du processus appelé transcription. L'activité d'un gène se laisse très bien lire à l'aide des molécules d'ARNm présentes dans la cellule. Toutefois, ces traces s'effacent rapidement : l'ARNm n'est pas stable ; la cellule dégrade souvent ces molécules après peu de temps.
L'anneau d'ADN comme système de stockage
Des chercheurs dirigés par Randall Platt du Département des systèmes biologiques de l'ETH Zurich à B?le ont maintenant mis au point un système de mémoire moléculaire qui enregistre les événements de transcription, et ce sous la forme d'une molécule d'ADN en forme d'anneau, appelée plasmide. Les informations stockées peuvent être lues en déterminant la séquence des éléments constitutifs de l'ADN du plasmide.
Pour leur "dispositif de stockage", les doctorants de Platt, Florian Schmidt et Mariia Cherepkova, ont eu recours au système Crispr/Cas. Celui-ci sert par exemple aux bactéries et aux archées de simple mémoire immunitaire, qui stocke des informations génétiques sur des agents pathogènes ayant déjà existé. L'enregistrement des informations a lieu à un endroit prédéfini du plasmide - le "Crispr-Array".
Enfiler l'information génétique comme des perles
Dans une telle matrice Crispr est déposé à chaque fois un court fragment d'ADN, appelé "spacer", qui provient de l'agent pathogène. Les spacers de différents agents pathogènes sont séparés les uns des autres par de courtes séquences d'ADN identiques, les "direct repeats", de sorte que l'ensemble ressemble à un collier de perles.
Les chercheurs ont introduit des gènes du système Crispr/Cas d'une autre espèce de bactérie dans le patrimoine génétique de la bactérie intestinale Escherichia coli ont été insérés dans les cellules. L'un de ces gènes Cas est lié à une transcriptase inverse, une enzyme qui, après avoir présenté des molécules d'ARN, produit des molécules d'ADN ayant le même contenu informatif, c'est-à-dire qu'elle retraduit l'information ARN en information ADN.
Complété par ces gènes étrangers Escherichia coli-Les cellules transcriptomiques ont été capables de produire un complexe de protéines moléculaires qui se lie à de courtes molécules d'ARNm. La transcriptase inverse traduit ces courtes molécules d'ARN en fragments d'ADN qui sponsorisent la même information que l'ARN original et qui sont ensuite intégrés dans la matrice CRISPR de manière analogue au processus naturel. Ce processus peut avoir lieu plusieurs fois et de nouveaux espaceurs sont insérés dans la matrice Crispr par ordre chronologique décroissant : Le dernier fragment d'ADN enregistré dans le temps est toujours placé en premier.
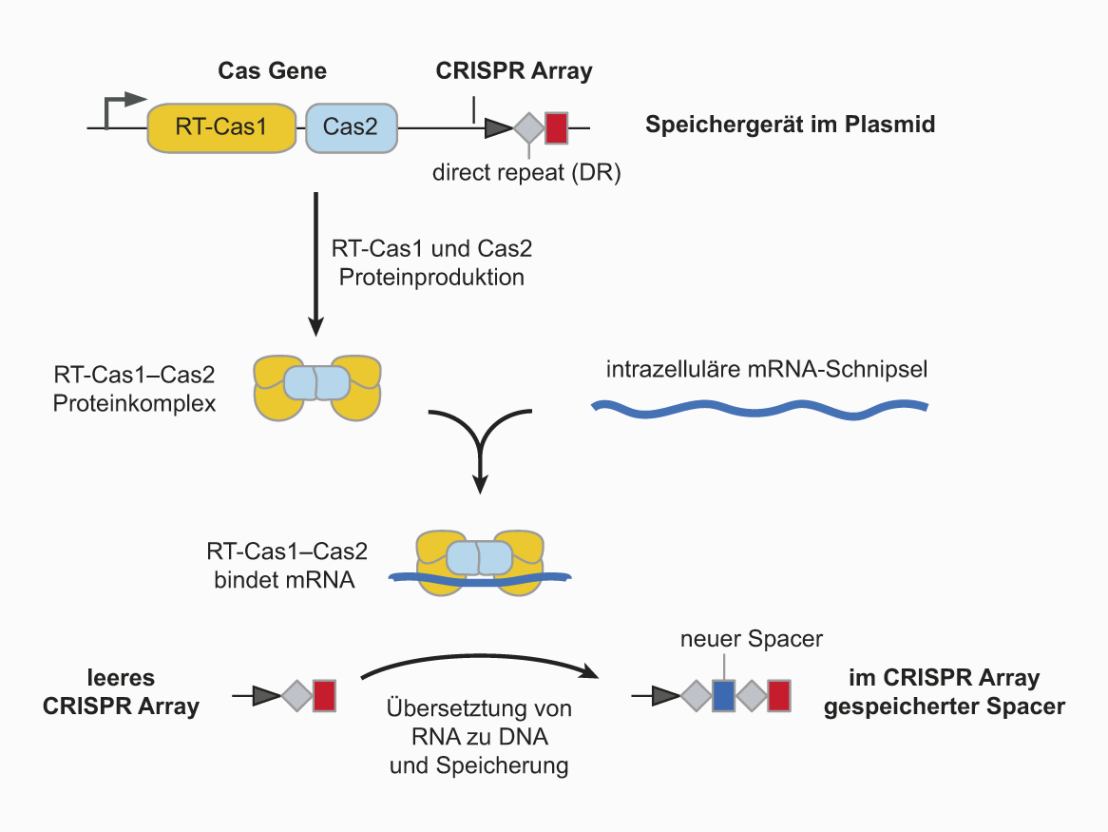
De cette manière, il est possible de stocker en soi autant de fragments d'ADN que l'on veut. L'ADN étant très stable, l'information qui y est stockée est conservée pendant longtemps et est en outre transmise d'une génération de bactéries à l'autre. En déterminant les éléments constitutifs de l'ADN dans les plasmides produits, les chercheurs peuvent finalement analyser quel gène a été actif, combien de fois et dans quel ordre chronologique.
"Ce système est un enregistreur de données biochimiques. Il nous permet d'enregistrer et de consulter sur plusieurs générations la réponse génétique des bactéries aux influences extérieures", explique Florian Schmidt, premier auteur de l'étude correspondante qui vient d'être publiée dans la revue spécialisée "Nature". "Cela fait longtemps que les chercheurs du monde entier travaillent sur de tels enregistreurs de données cellulaires artificiels. Mais nous sommes sans doute les premiers à en avoir développé un capable d'enregistrer une telle diversité de signaux", déclare non sans fierté le professeur Platt de l'ETH. Les chercheurs b?lois ont travaillé plus de deux ans sur ce système.
Exploiter le journal de bord complet
Jusqu'à présent, les chercheurs devaient souvent se contenter d'un instantané de la vie d'une cellule. Pour enregistrer cette image, ils doivent généralement détruire les cellules, extraire l'ARNm et le quantifier à grands frais. Dans le réseau Crispr, les cellules enregistrent toutefois une évolution sur plusieurs stades différents, ce qui permet aux chercheurs d'évaluer quasiment tout un journal de bord.
Dans le cadre de leur étude, les chercheurs de l'EPFZ ont montré comment des cellules de cellules souches équipées d'un enregistreur de données pouvaient être utilisées pour le stockage de données. E.-coli-Les chercheurs ont découvert que les bactéries réagissent à l'herbicide paraquat. Le poison a déclenché une réponse génétique dans les cellules, que les scientifiques pouvaient encore lire des jours plus tard sur la matrice Crispr. Sans l'enregistreur de données, les traces moléculaires indiquant le contact avec l'herbicide se seraient alors dégradées depuis longtemps et l'information aurait été perdue.
L'enregistreur biochimique pourrait être utilisé en premier lieu dans la recherche, mais il est également envisageable de l'utiliser comme une sorte de capteur pour mesurer les toxines environnementales, comme dans le cas de l'herbicide, ou dans le domaine du diagnostic. Le chemin vers une application pratique est toutefois encore long. La présente étude n'est qu'une preuve de faisabilité. Les chercheurs de Randall Platt s'efforcent actuellement d'appliquer le système à d'autres types de cellules afin d'ouvrir la voie à des outils utilisables pour le diagnostic.
Référence bibliographique
Schmidt F, Cherepkova MY, Platt RJ. Enregistrement transcriptionnel par acquisition d'espaceur CRISPR à partir de l'ARN. Nature, 3 octobre 2018, doi : page externe10.1038/s41586-018-0569-1