Cosa rivela il nuovo pan-genoma sui geni dei bovini
I ricercatori dell'ETH di Zurigo hanno confrontato i genomi di riferimento di diverse razze bovine domestiche e di bovini selvatici strettamente imparentati. Hanno scoperto geni con funzioni precedentemente sconosciute.

La ricerca genetica di oggi lavora spesso con i cosiddetti genomi di riferimento. Si tratta di dati provenienti da sequenze di DNA che gli scienziati hanno raccolto come esempio rappresentativo del patrimonio genetico di una specie.
Per creare un genoma di riferimento, i ricercatori di solito utilizzano sequenze di DNA provenienti da uno o pochi individui, che rappresentano male la diversità genetica complessiva degli individui o delle sottopopolazioni. Pertanto, le referenze non sempre corrispondono esattamente al set di geni di un particolare individuo.
Poiché fino a pochi anni fa era molto complesso, costoso e lungo produrre tali genomi di riferimento, la ricerca si è concentrata sui genomi degli esseri umani e dei più importanti organismi biologici modello, come il verme del filo. C.elegans.
Tuttavia, oggi i ricercatori hanno deciso di utilizzare macchine di sequenziamento veloci, algoritmi sofisticati che assemblano le sequenze di DNA in interi cromosomi e molta più potenza di calcolo, per cui i genomi di riferimento vengono creati sempre più spesso anche per altre specie. Se i ricercatori vogliono capire meglio l'evoluzione e altre questioni fondamentali della biologia, hanno bisogno di genomi di riferimento di alta qualità provenienti dal maggior numero possibile di specie.
Questo vale anche per gli animali da allevamento. Fino a poco tempo fa, per i bovini domestici (Bos taurus) era disponibile un solo genoma di riferimento: quello della vacca "Dominette" della razza "Hereford". I ricercatori hanno utilizzato questa referenza per confrontare altre sequenze di DNA di bovini al fine di individuare le variazioni genetiche e definire i genotipi corrispondenti. Tuttavia, le referenze precedenti non riflettono la diversità della specie, poiché non contengono varianti genetiche che differenziano gli individui.
Lacuna colmata
Un team di ricerca guidato da Hubert Pausch, professore di genomica animale all'ETH di Zurigo, ha ora colmato questa lacuna: I ricercatori hanno creato un cosiddetto pangenoma utilizzando i genomi di altre tre razze bovine domestiche, tra cui l'originaria razza bruna svizzera, due specie strettamente imparentate come il bovino zebù e lo yak, nonché il precedente genoma di riferimento del bovino domestico. Lo studio corrispondente è stato appena presentato sulla rivista scientifica PNAS.
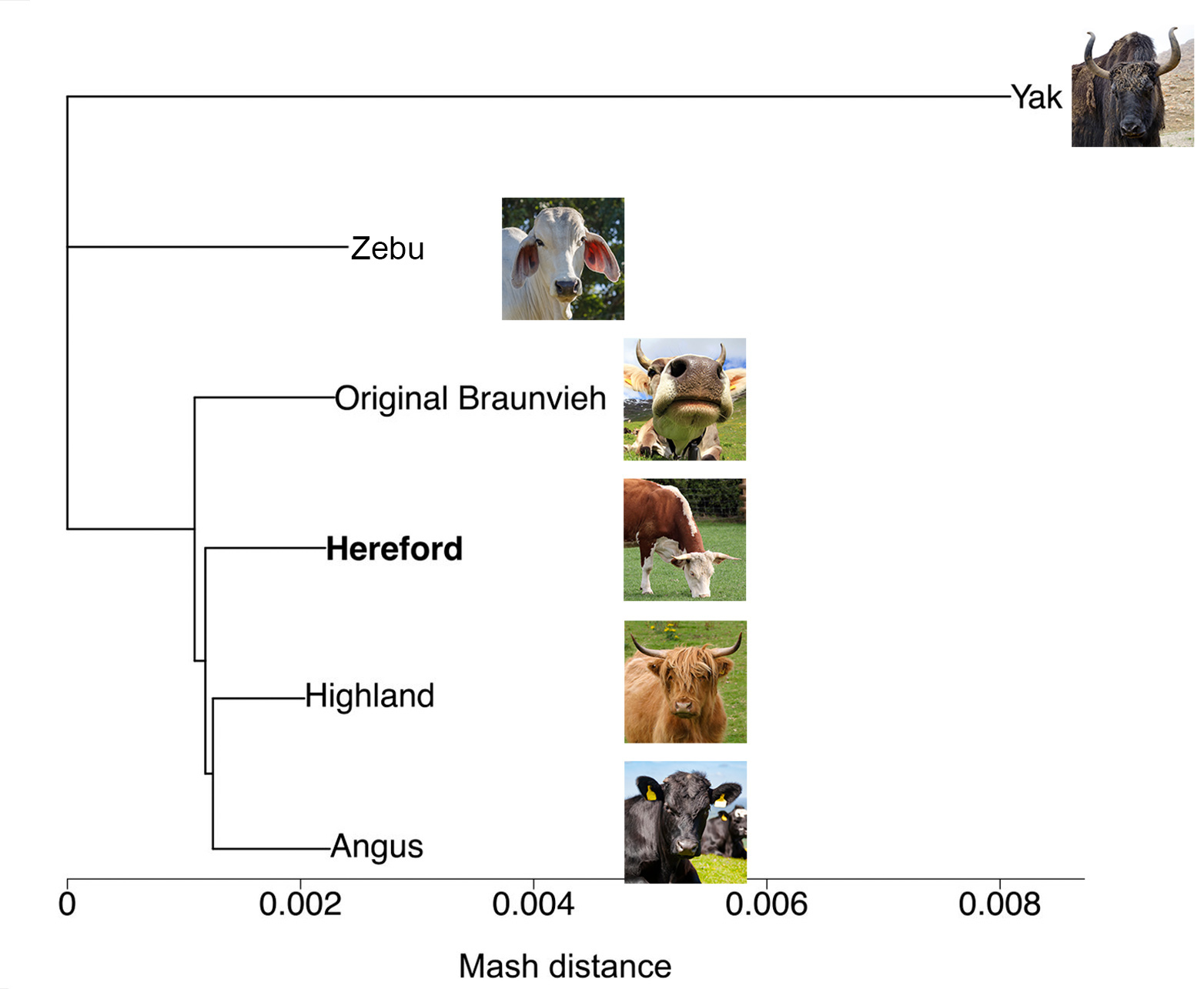
Questo pangenoma bovino integra le sequenze contenute nei sei genomi di riferimento individuali. "In questo modo, siamo stati in grado di mostrare con estrema precisione quali sequenze mancano, ad esempio, nel genoma di riferimento dell'Hereford, ma sono presenti nel nostro genoma originale di Braunvieh o nei genomi di altre razze e specie bovine", spiega Pausch.
Trovati nuovi geni e funzionalità
I ricercatori dell'ETH hanno trovato numerose sequenze di DNA e persino interi geni che mancavano nel precedente genoma di riferimento della mucca Hereford. Analizzando i trascritti di questi geni (cioè le molecole di RNA messaggero) in un'ulteriore fase, i ricercatori sono stati in grado di classificare alcune delle nuove sequenze trovate come funzionali e biologicamente rilevanti. Ad esempio, molti dei geni trovati sono associati a funzioni immunitarie: Negli animali che avevano avuto contatto con batteri patogeni, questi geni erano più o meno attivi rispetto a quelli che non avevano avuto contatto con gli agenti patogeni.
Questo lavoro è stato reso possibile da una nuova tecnologia di sequenziamento disponibile da un anno presso il Centro di Genomica Funzionale dell'ETH di Zurigo. Con questa nuova tecnologia, i ricercatori possono leggere con precisione lunghe sezioni di DNA, rendendo meno complesso il processo computazionale per assemblare correttamente le sezioni analizzate. "La nuova tecnologia ha semplificato il processo di assemblaggio di un genoma. Ora possiamo creare genomi di riferimento da zero in modo rapido e accurato", afferma Pausch. Inoltre, il costo di tali analisi è diminuito, il che significa che i ricercatori possono ora generare genomi di qualità di riferimento da molti individui di una specie.
I ricercatori dell'ETH lavorano a stretto contatto con il consorzio "Bovine Pangenome". L'obiettivo è quello di creare un genoma di riferimento di almeno un animale di ciascuna razza bovina del mondo. Anche il materiale genetico dei parenti non addomesticati dei bovini domestici sarà analizzato in questo modo.
Possibile un allevamento più mirato
Il consorzio e l'ETH Pausch sperano che la collezione di genomi di riferimento consenta di trovare varianti geniche che non esistono più negli animali domestici, ma che esistono nei parenti selvatici. Ciò fornirà indizi su quali tratti genetici sono stati persi con l'addomesticamento.
"Diventa particolarmente interessante quando si confrontano i nostri bovini autoctoni con gli zebù o con razze adattate ad altre condizioni climatiche", spiega l'esperto. In questo modo la ricerca potrà sapere quali varianti genetiche rendono gli animali degli ambienti tropicali più resistenti al calore. Il passo successivo potrebbe essere quello di incrociare specificamente queste varianti in altre razze bovine o di introdurle proprio attraverso l'editing del genoma. Tuttavia, la strada da percorrere è ancora lunga. Il nuovo pangenoma bovino consente ora ai ricercatori di individuare in modo più rapido e preciso i geni e le varianti del DNA che differiscono tra le razze bovine.
Letteratura di riferimento
Crysnanto D, Leonard AS, Fang Z-H, Pausch H. Nuove sequenze funzionali scoperte attraverso un grafo multiassemblaggio di bovini. PNAS, 18 maggio 2021, 118 (20) e2101056118; doi: pagina esterna10.1073/pnas.2101056118
150 anni di scienze agrarie

Il Dipartimento amministrativo è stato fondato all'ETH di Zurigo nel 1871. Ora l'Istituto di scienze agrarie celebra il suo 150° anniversario con vari eventi nell'anno del giubileo.
Sul sito Sito web dell'anniversario troverete una timeline con fatti storici e articoli selezionati degli ultimi 150 anni.