Il metodo Fleming in miniatura
I ricercatori del Dipartimento biosistemi e ingegneria dell'ETH di Zurigo a Basilea hanno sviluppato un metodo che consente di testare rapidamente l'effetto antibiotico di un numero molto elevato di molecole. Hanno così scoperto nuovi candidati per gli antibiotici. In futuro, vorrebbero utilizzare il metodo anche per analizzare campioni di terreno e il microbioma della pelle umana alla ricerca di organismi interessanti dal punto di vista medico.
90 anni fa, una muffa finì involontariamente su una coltura batterica nel laboratorio di Alexander Fleming. Il batteriologo scozzese osservò che il fungo produceva una sostanza che uccideva i batteri sulla piastra della coltura cellulare. Scoprì così la penicillina, uno dei primi antibiotici. Oggi esistono diverse decine di classi di antibiotici e la scienza continua a cercare instancabilmente nuovi agenti antimicrobici perché sono urgentemente necessari in medicina. La maggior parte di questi farmaci sono sostanze naturali o si basano su di esse. E il metodo di rilevamento è ancora lo stesso dei tempi di Fleming: Un antibiotico è una sostanza in grado di uccidere i batteri su una piastra di coltura cellulare.
Gli scienziati guidati da Steven Schmitt del gruppo del professor Sven Panke dell'ETH di Zurigo, presso il Dipartimento di Scienza e Ingegneria dei Biosistemi di Basilea, hanno ora modernizzato e miniaturizzato il metodo di Fleming, rendendolo adatto all'analisi high-throughput dei microrganismi e dei principi attivi da essi prodotti. "Mentre attualmente ci vuole fino a un anno per analizzare 10.000 produttori di principi attivi con i metodi convenzionali, noi possiamo analizzare milioni di varianti in pochi giorni", spiega Schmitt, che ha sviluppato il metodo nell'ambito della sua tesi di dottorato all'ETH.
Utilizzando la nuova tecnologia, gli scienziati dell'ETH, insieme a colleghi olandesi e tedeschi, sono riusciti a identificare una serie di nuovi candidati antibiotici. In una fase successiva, i ricercatori studieranno se questi candidati includono anche quelli adatti all'uso in medicina.
"Tè alle bollicine per la scoperta di farmaci
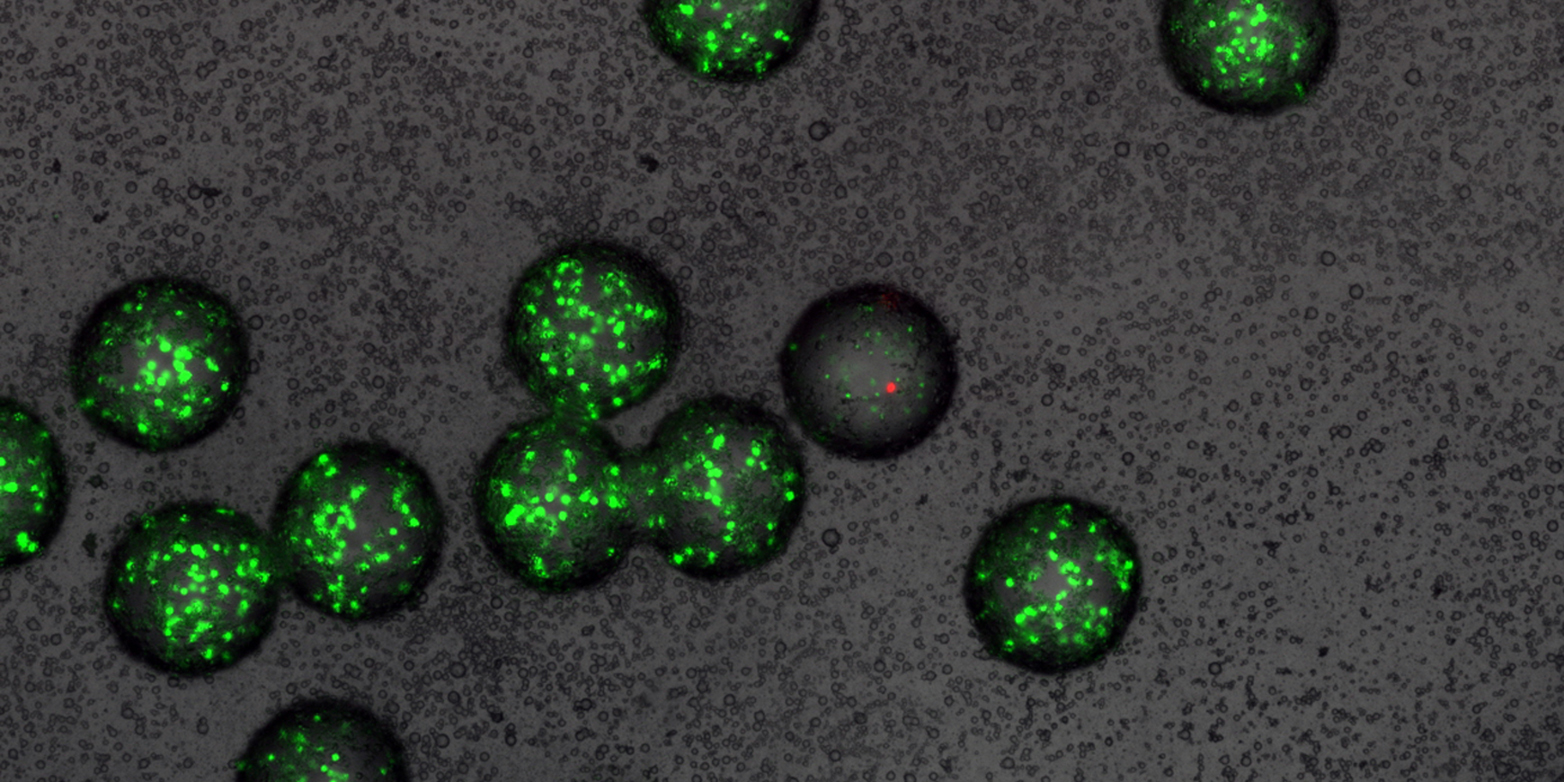
Ciò che Alexander Fleming faceva con un piatto di coltura cellulare di dieci centimetri di diametro, la nuova tecnica chiamata "Nano-Fleming" utilizza piccole perle di gel di appena mezzo millimetro - simili al "bubble tea" o all'imitazione del caviale della cucina molecolare. Gli scienziati possono utilizzare queste perle per testare l'effetto antibiotico di nuove sostanze. A tal fine, da un lato inseriscono nelle perle diversi batteri sensori e dall'altro un microrganismo per perlina, producendo così una sostanza potenzialmente antibiotica da testare.
Se la sostanza prodotta ha effettivamente un effetto antibiotico, i batteri sensori muoiono. Se non è efficace, si moltiplicano e formano ammassi di cellule. Dopo aver etichettato i batteri sensori con un colorante fluorescente, gli scienziati possono selezionare le microsfere di gel debolmente fluorescenti utilizzando un metodo di selezione ad alto rendimento. Queste contengono un microrganismo che ha prodotto un antibiotico efficace. Gli scienziati possono quindi identificare questa sostanza attiva.
Antibiotici più efficaci
Nello studio attuale, che i ricercatori hanno pubblicato sulla rivista pagina esternaNature Chemical Biology hanno testato una collezione di 6000 peptidi (brevi proteine) per verificarne l'effetto antibiotico. Si trattava di molecole simili a un gruppo di antibiotici peptidici noti come lantibiotici. Gli scienziati volevano verificare se fosse possibile aumentarne l'efficacia o aggirare i meccanismi di resistenza noti modificando in modo intelligente la struttura molecolare dei lantibiotici.
Insieme ai colleghi olandesi e tedeschi, sono partiti dai lantibiotici noti e dalle loro subunità strutturali e funzionali. Utilizzando un approccio biotecnologico, hanno combinato le subunità di vari lantibiotici in tutti i modi possibili, creando così una collezione di microrganismi che producono questi nuovi peptidi combinati. Hanno poi testato questi microrganismi con il metodo del "nano-Fleming". In effetti, hanno trovato undici peptidi che, rispetto ai lantibiotici conosciuti, funzionano in dosi minori o sono in grado di superare i meccanismi di resistenza noti.
Ricerca in natura
"Il metodo è anche un ottimo modo per verificare se i microrganismi presenti in natura producono sostanze attive non ancora scoperte", spiega Schmitt. Il fatto che i microbi cerchino di eliminare i loro concorrenti con composti biochimici è un meccanismo naturale e diffuso. ? quindi possibile che nuove classi di antibiotici si trovino in habitat come i campioni di suolo o il microbioma ancora poco studiato della pelle umana e della saliva. La nuova tecnologia potrebbe essere utilizzata per analizzare molto bene i microrganismi di questi ambienti. "E poiché ora possiamo testare molti più produttori di sostanze attive in un breve lasso di tempo rispetto ai metodi precedenti, le possibilità di scoprire sostanze attive da microrganismi rari aumentano".
La tecnica potrebbe essere adattata anche per testare altri criteri per gli antibiotici durante l'analisi iniziale, come la stabilità nel flusso sanguigno umano o l'elusione dei meccanismi di resistenza. Oppure sarebbe possibile equipaggiare le sfere di gel con diversi batteri sensori: quelli che un principio attivo deve assolutamente uccidere, ad esempio gli agenti patogeni, e altri che non deve danneggiare in nessun caso, ad esempio i batteri della pelle sana o della flora orale.
Schmitt sta ora pianificando ulteriori sviluppi come questi nell'ambito dell'ETH Pioneer Fellowship e di una borsa di studio ponte, con la quale il Fondo Nazionale Svizzero per la Ricerca Scientifica e Innosuisse promuovono congiuntamente gli sviluppi all'interfaccia tra scuole universitarie e industria. ? prevista anche la fondazione di un'apertura per la commercializzazione del metodo.
Riferimento alla letteratura
Schmitt S, Montalbán-López M, Peterhoff D, Deng J, Wagner R, Held M, Kuipers OP, Panke S: Analisi di lantineptidi antimicrobici modulari bioingegnerizzati su scala nanolitrica. Nature Chemical Biology 2019, 15: 437, doi: pagina esterna10.1038/s41589-019-0250-5pagina esterna